適応回路を規定する1細胞空間エピゲノムセンサス技術の確立 /Development of single-cell spatial epigenomics technology for adaptive circuit census

小松 哲郎 /Tetsuro Komatsu
群馬大学 生体調節研究所 講師
Gunma University, Institute for Molecular and Cellular Regulation, Lecturer
HPリンク
研究室HP:https://epigenetics.imcr.gunma-u.ac.jp/
リサーチマップ:https://researchmap.jp/t_komatsu
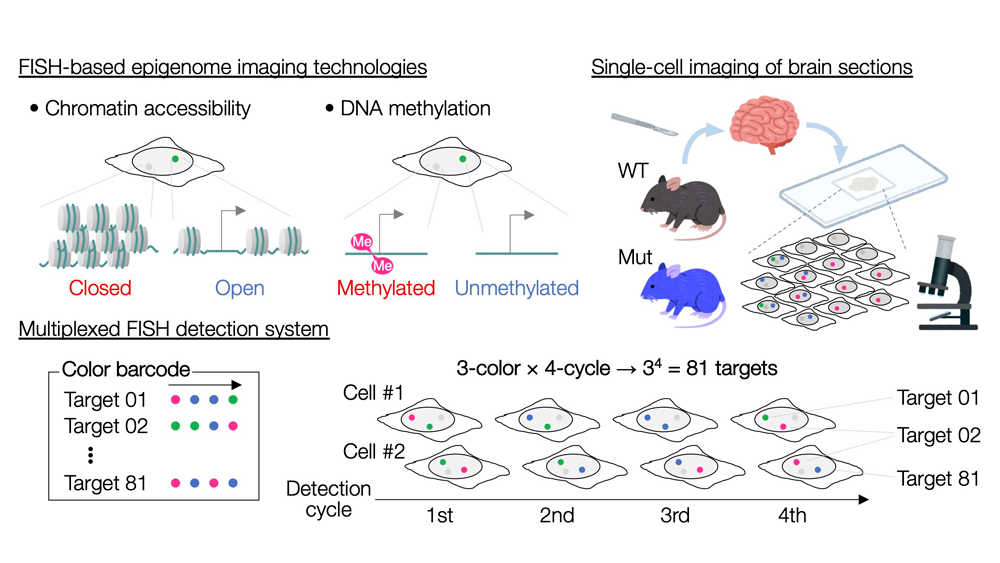
動物の行動適応の基盤となる、神経細胞ネットワークの構築機構や動的性質の理解には、1細胞解像度での解析が必須である。しかし、1細胞シークエンシング解析では、細胞の分離・可溶化に伴い、細胞の形態や組織内局在といった「空間情報」が失われる。そのため、空間情報を保持したままイメージングにより網羅的解析を行う空間オミクス技術が、現在注目を集めている。例えば、蛍光in situハイブリダイゼーション(FISH)法を基盤としたトランスクリプトーム解析技術は、転写産物であるmRNAを網羅的に検出することで、細胞ごとの遺伝子発現プロファイリングを可能とする。一方、遺伝子のON/OFFを決定する上流の分子機構、すなわち発現するべき遺伝子の選択を担う転写制御因子のゲノム結合には、クロマチンの化学修飾やアクセシビリティといったエピゲノム制御が重要な役割を果たす。しかし、エピゲノムを1細胞レベル且つ高空間解像度で解析する技術は未だ限られており、神経回路の構築・遷移を規定するエピゲノム制御機構の詳細は、未だ不明である。
我々は、蛍光イメージング解析と次世代シークエンサーを用いたオミクス解析を両軸とした研究を展開している。上記背景の下、近年は特にFISH法を応用した空間エピゲノム解析技術の開発を推進している。本研究では、クロマチンアクセシビリティやDNAメチル化といった、ゲノム領域特異的なエピゲノム状態をFISHにより可視化する独自イメージング技術をマウス脳組織切片に適用し、適応脳機能を担うゲノム領域及びそのエピゲノム制御の、1細胞解像度での包括的理解に挑む。
文献
- Schwartz U, Komatsu T et al. Changes in adenoviral chromatin organization precede early gene activation. EMBO J. 2023 Oct 4;42(19):e114162.
- Suzuki T†, Komatsu T†, Shibata H† et al. Crucial role of iron in epigenetic rewriting during adipocyte differentiation mediated by JMJD1A and TET2 activity. Nucleic Acids Res. 2023 Jul 7;51(12):6120-6142. (†Equal contribution)
- Suzuki T, Hayashi M, Komatsu T et al. Measurement of the nuclear concentration of α-ketoglutarate during adipocyte differentiation by using a fluorescence resonance energy transfer-based biosensor with nuclear localization signals. Endocr J. 2021 Dec 28;68(12):1429-1438.
- Komatsu T et al. In vivo labelling of adenovirus DNA identifies chromatin anchoring and biphasic genome replication. J Virol. 2018 Sep;92(18):e00795-18.



